Metodología del modelado
Estrategia 1
El objetivo inicial de este trabajo era realizar un modelo del precursor de la proteína de unión a la vitamina D (DBP). Para ello había que encontrar primero los templates apropiados.
Como miembro de la familia de las albúminas plasmáticas, la DBP comparte una serie de característica con el resto de proteínas como el patrón repetitivo de puentes disulfuro y la estructura tridimensional de 3 dominios divididos a su vez en subdominios.
Por ello, a la hora de modelar la proteína humana de unión a la vitamina D se esperaba utilizar como template algún miembro de su familia.Se hizo una primera búsqueda mediante el programa BLAST en la base de datos del SWISSPROT y se generó una matriz para posteriormente utilizarla en una segunda búsqueda contra la base de datos del PDB (tabla 1).
Score E Sequences producing significant alignments: (bits) Value
Sequences used in model and found again:/seq/databases/pdb/scratch/pdb1bj5.ent Chain 642 0.0
/seq/databases/pdb/scratch/pdb1e7c.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1e7e.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1e7f.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1e7g.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1e7i.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1gni.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1gnj.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1h9z.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1ha2.ent Chain A 642 0.0
/seq/databases/pdb/scratch/pdb1e7h.ent Chain A 641 0.0
/seq/databases/pdb/scratch/pdb1bke.ent Chain 639 0.0
/seq/databases/pdb/scratch/pdb1uor.ent Chain 638 0.0
/seq/databases/pdb/scratch/pdb1ao6.ent Chain A 637 0.0
/seq/databases/pdb/scratch/pdb1ao6.ent Chain B 637 0.0
/seq/databases/pdb/scratch/pdb1bm0.ent Chain A 637 0.0
/seq/databases/pdb/scratch/pdb1bm0.ent Chain B 637 0.0
/seq/databases/pdb/scratch/pdb1e78.ent Chain A 637 0.0
/seq/databases/pdb/scratch/pdb1e78.ent Chain B 637 0.0
/seq/databases/pdb/scratch/pdb1e7a.ent Chain A 637 0.0
/seq/databases/pdb/scratch/pdb1e7a.ent Chain B 637 0.0
/seq/databases/pdb/scratch/pdb1e7b.ent Chain A 637 0.0
/seq/databases/pdb/scratch/pdb1e7b.ent Chain B 637 0.0CONVERGED!
Tabla 1. Resultados de búsqueda en PDB
Aparecen como proteínas homólogas las albúminas, miembros también de la misma familia. Se pensó que podían ser un buen template debido a su estructura similar formada por tres dominios.
Pero el hecho de que el porcentaje de identidad era bastante bajo (21%) y que estudios recientes demuestran que aunque tienen similar topología, existen diferencias importantes en el plegamiento general y local de ambas proteínas llevó a comprobar primero las diferencias estructurales entre ambas antes de utilizar las albúminas como template para crear un modelo de la DBP.
Comparación estructural albúmina - DBP:
En primer lugar se realizó un alineamiento estructural mediante el programa STAMP y una posterior visualización mediante el programa RASMOL, y los resultados obtenidos no fueron muy favorables; como se esperaba, la orientación global de cada dominio es muy diferente, lo que resulta en dos estructuras totalmente distintas. El cálculo mediante el programa XAM de las desviaciones rms entre carbonos a del primer dominio es de 3.3 Å.
Los valores de RMSDs general y por dominios (Verboven et al. 2002) demuestran numéricamente lo ya observado :rmsd general : 10.1 Å
rmsd dominio I : 4.01 Å
rmsd dominio II : 4.2 Å
rmsd dominio III : 3.9 ÅEstos resultados llevaron a plantearse cómo dos proteínas de la misma familia, conservando un mismo patrón de estructural de dominios y subdominios y realizando una misma función de transporte plasmático, podían haber adoptado un plegamiento diferente. La respuesta quizás podría estar en la especialización de función que han ido sufriendo estas proteínas.
La unión de la DBP a la vitamina D y a sus metabolitos se realiza por una región del dominio I (hélices 1-4). El estudio estructural comparativo del dominio I de la albúmina y DBP puede explicar porqué la vitamina D sólo puede unirse a DBP.
Así, la superposición del dominio I de la albúmina y de DBP muestra que la hélice 4 de la albúmina forma un contacto próximo con las hélices 2 y 3, no dejando sitio para la vitamina D (Fig4).
También se comprobó si los aminoácidos de la DBP implicados en la unión a la vitamina D estaban conservados en la albúmina. Se vio que los aminoácidos polares implicados en la formación de puentes de hidrógeno con la vitamina D han sido sustituidos por aminoácidos hidrofóbicos. De esta manera, la Tirosina 32 y la Metionina 107 son en la albúmina fenilalanina. La Serina 76 ha cambiado a glicina.
Los aminoácidos aromáticos presentes en la DBP y que hacen stacking con la vitamina D, como son la Phe 36 y la Tyr 68 también han sido sustituidos por otros no aromáticos pero manteniendo su hoidrofobicidad. En cambio, los aminoácidos hidrofóbicos están conservados. Este hecho puede explicarse debido a la función de transporte de ácidos grasos y otros ligandos hidrofóbicos que realiza la albúmina; para poder unirse a ellos necesita aminoácidos hidrofóbicos.
Fig 4. Superposición subdominio Ia de albúmina (azul) y DBP (rojo)Se observó tambien que el Trp-145 anteriormente citado, esencial para la unión de la vitamina D, no está conservado en la albúmina, por lo que cabe preguntarse si una mutación en la posición 145 separó a la DBP de la albúmina, convirtiéndose DBP en un transportador específico de vitamina D (Swamy et al. 1995).Pero quizás, las diferencias estructurales más importante entre albúmina y DBP, se deban a la capacidad de esta última de unirse a filamentos de actina. Esta unión involucra los tres dominios de la DBP que forman una gran cavidad donde se asientan los polímeros de actina, cavidad que no se observa en la albúmina (Otterbein et al. 2002).
Por tanto, parece razonable proponer que las diferencias estructurales entre albúmina y DBP están directamente relacionadas con la unión de la DBP a la actina, que involucra los tres dominios, más que con la capacidad específica de la DBP de unirse a vitamina D que tan solo involucra las 4 primeras hélices del dominio I.
Comparación estructural VDR-DBP:
Se ha visto que los efectos de la vitamina D y sus análogos están mediados por la unión a un receptor intracelular de la vitamina D (VDR) (Ray, 1996). Basándose en este hecho, se comparó estructuralmente la DBP con la VDR, para estudiar posibles analogías, pero la superposición estructural muestra grandes diferencias (Fig 5). Mientras que en VDR el sitio de unión a la vitamina D es un bolsillo cerrado en el interior del receptor, en DBP es una grieta localizada en la superficie de la molécula y, en parte, en contacto con el solvente.
Por tanto, tampoco sería un buen template para modelar la vitamina D.
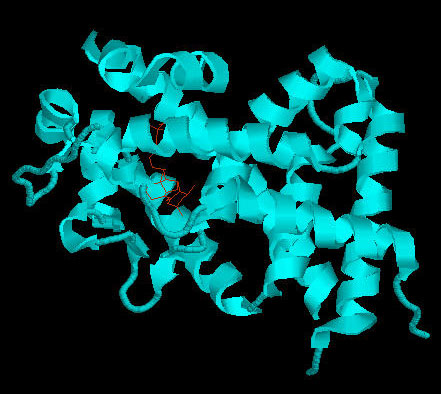
Fig 5. VDR unida a vitamina D
Home | Abstract | Características estructurales | Metodología 2 | Conclusiones | Bibliografía