 SUS
AMIGUETES.
SUS
AMIGUETES. SUS
AMIGUETES.
SUS
AMIGUETES.
El primer lugar donde buscar fue la base de datos Swissprot.Allí
encontraríamos, con ayuda de Psi-blast (programa que busca
secuencias homólogas de forma iterativa hasta que llega a un punto
en que ya no encuentra más proteínas relacionadas)
otras proteínas homólogas a Floripondio ordenadas segun su
e-value y su score. Los primeros amigos que hizo nuestra proteína,
tras llegar a la convergencia, fueron los siguientes:
*
EN PRIMERA POSICION Y CON UN E-VALUE DE 3* e-39... HIT_BACSU
HIT PROTEIN (O07513)
*Siguiendolo
de muy cerca, con un e-value de 2 * e-38...YHIT_MYCLE HYPOTHETICAL
HIT-LIKE PROTEIN U296A ( P49774)
*
En tercera posición, con un e-value de 3 * e-37... GAL7_ECOLI
GALACTOSE-1-PHOSPHATE URIDYLYLTRANSFERASE (P09148)
*
Y cerrando la lista de amiguetes selecionados, con un e-value de 1 * e-36...HNT1_YEAST
HIT FAMILY PROTEIN 1 (Q04344)
Con las secuencias en formato fasta de estas secuencias, el programa CLUSTALW nos creará un alineamiento múltiple de las secuencias de estas proteínas (sw1.aln) que será el que el programa HMMER utilizará para darnos la matriz oculta de Markov (mod1.hmm) , que nos da un perfil secuencial de nuestra proteina. Esta matriz se pasará de nuevo a formato clustalw para utilizarlo como input en el Psi-blast y así buscar en PDB estructuras homólogas.
Había muchísimos amiguetes de estructura similar. Nos dimos cuenta de que todos los amiguetes de floripondio pertenecían a una misma super-família de proteínas, las HIT proteins.
Para escoger a los mejores amigos de floripondio, seleccionamos algunos en función de su e-value. Se consultó la base de datos del SCOP para seleccionar templates de distintos organismos para no sesgar la muestra de amiguetes escogiendo dos templates casi idénticos (dos proteínas de un mismo organismo y con una misma función que pertenezcan a la misma família estructural, seguramente se habrán generado por duplicación y no habrán tenido tiempo suficiente para divergir).
Los afortunados fueron:
* Del Caenorhabditis elegans (nématodo) nos quedamos con:
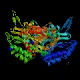 pdb1ems.ent
(esta secuencia dará mucho que hablar en capítulos posteriores).
Tiene une-value de 2e
-40. Se trata de un homodímero
con dos subunidades iguales en cada cadena.
pdb1ems.ent
(esta secuencia dará mucho que hablar en capítulos posteriores).
Tiene une-value de 2e
-40. Se trata de un homodímero
con dos subunidades iguales en cada cadena.
* Del Oryctolagus
cuniculus (de un conejo, vaya):
El primero estaba acomplejado con adenosina y el segundo se encuentra solo. Ambos tienen un e-value de 3e -36.
* Del Homo sapiens (uno de los nuestros):
Ambas son de la família de las FHIT (fragile histidine triade related protein), una rama de la superfamília HIT. La primera de ellas se encuentra acomplejada con un análogo de AMP, mostrando un e-value de 1e -38. La segunda está más solita que la una y tiene un e-value de 5e -36.
Tienen el mismo e-value los dos (6e -38) y pertenecen a la familia
de inhibidores de la PKC (la otra rama de l super-família),
siendo el segundo de ellos el que se encuentra acomplejado. Ambos son homodímeros,
por lo que tendrán dos cadenas: la A y la B.
De los tres amiguetes que tienen dos cadenas, debemos escoger una de ellas: en los tres casos elegimos la cadena A por aparecer antes en los resultados del Psi-blast (¿quién dijo que los últimos serán los primeros?)
Así pues, contamos con 7 templates de proteínas pertenencientes
a la
super-família
de las HIT proteins que Floripondio para
poder elucidar su estructura tridimensional.