$ cd practica_1
$ cd PDB (hi ha una llarga llista de pdb que usarem per treballar amb el rasmol)
$ emacs pdb1ay1.ent & (si obrim amb un editor un d'aquests pdb se'ns visualitza un document com aquest)
ATOM 1 N ASP L A 1 27.504 -17.288 18.937 ATOM 2 CA ASP L A 1 27.334 -16.029 19.656 ATOM 3 C ASP L A 1 27.777 -14.855 18.795 ATOM 4 O ASP L A 1 28.548 -13.993 19.232 ATOM 5 CB ASP L A 1 28.128 -16.048 20.959 ATOM 6 CG ASP L A 1 27.644 -15.011 21.948 ATOM 7 OD1 ASP L A 1 26.551 -14.437 21.738 ATOM 8 OD2 ASP L A 1 28.353 -14.782 22.949 ...
Aquesta llista és tant llarga com àtoms té la molècula. El primer terme ens indica si és un àtom (ATOM), un heteroàtom (HETATM) o si trobem TER voldrà dir que la llista anterior ha finalitzat. La segona columna enumera els àtoms. La tercera ens diu la naturalesa de cada un d'ells (N: nitrogen, CA: carboni alfa, C: carboni, O: oxigen). A continuació ens diu de quin aminoàcid es tracta (en aquest cas asparagina). La següent columna es refereix a la cadena a la qual pertany aquell aa (en aquest cas A), si és que la molècula està formada per més d'una cadena. Les següents tres columnes són les coordenades cartasianes de l'àtom en l'espai. I finalment, en aquest pdb en concret no apareix però es pot donar el cas que hi hagi dues columnes més: una referent a la ocupancia i l'altre al B-factor (que ens dóna una idea de la flexibilitat de la molècula).
$ rasmol pdb1an2.ent (l’ordre és aquesta si prèviament hem fet un “alias” o si estem treballant en tcsh)
Abans d’entrar a utilitzar cap comanda analitzem la informació que ens aporta la finestra del shell:
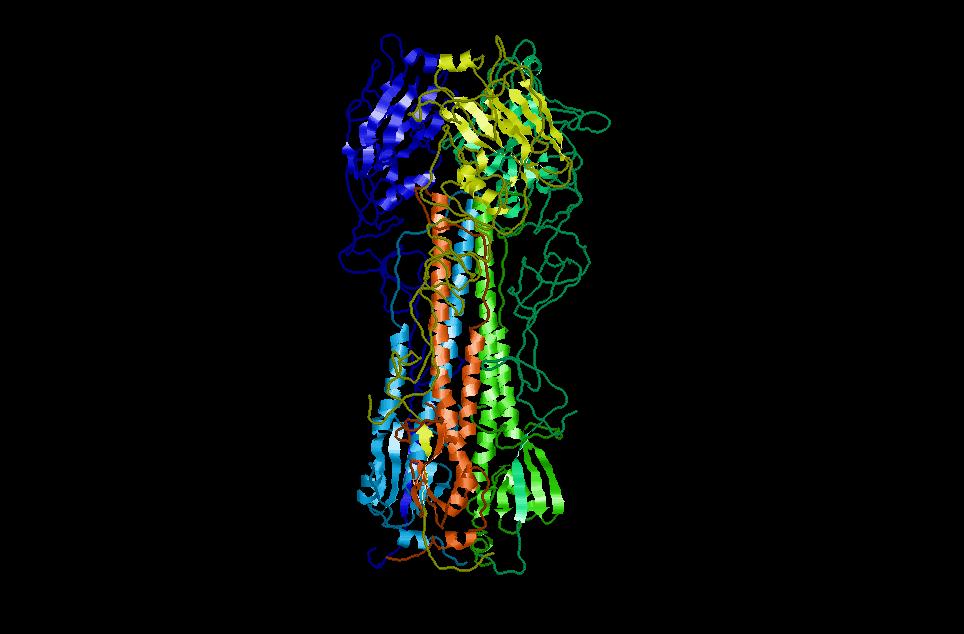
Molecule name..........HEMOGLOBIN I (HOMODIMER) Classification.........OXYGEN TRANSPORT Secondary Structure....PDB Data Records Database Code..........3SDH Number of Chains.......5 Number of Groups.......290 (267) Number of Atoms........2304 (355) Number of Helices......16 Number of Strands......0 Number of Turns........0 Number of Bonds........2435
Visualitzem la estructura per pantalla i la podem modificar amb les
següents comandes, que escrivim a la pantalla del shell:
rasMol> color chain (es veu una cadena de cada color)
rasMol> background red (si el que vols és que el color de la pantalla, que per defecte sempre és negre, sigui vermell)
rasMol> hbonds (et mostra els ponts d'hidrogen)
rasMol> ssbonds (et mostra els ponts dissulfur)
rasMol> select *D (selecciona la cadena D i les ordres que vinguin a
continuació només s'executaran sobre aquesta cadena)

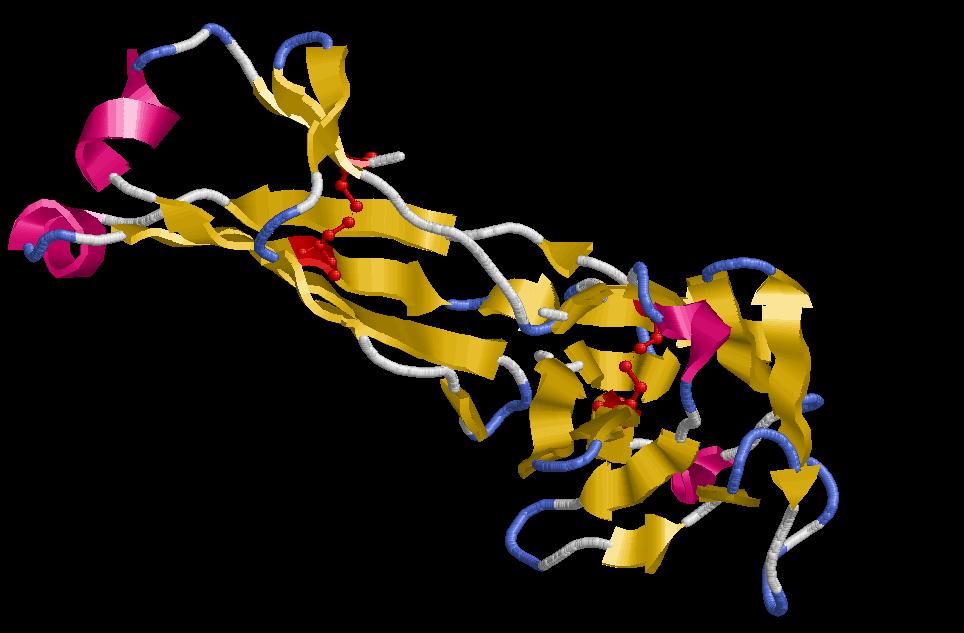
rasMol> select polar (seleccionem els residus polars, com es veu a la fotografia marcat en groc. Aquesta fotografia està feta també per veure l'angle que fan els residus amb la vertical de la hèlix, que és el mateix per a tots)
rasMol> select hydrophobic (selecciones els residus hidrofòbics)
rasMol> select 188 (seleccionem un residu concret, el 188; cada vegada que selecciones un residu t’informa del número d’àtoms que implica)
rasMol> select 188-190 (selecciones del residu 188 al 190)
rasMol> color magenta (ara només ens apareixerien liles els tres residus que hem seleccionat en la ordre anterior)
Per obtenir la imatge hem fet: rasMol> select 20, 24, 28, 32, 36, 40, 44, 48 (seleccionem cada 4 aminoàcids per comprovar que es tracta d’una hèlix, que fa un gir cada 4 residus)
rasMol> pick distance ( a continuació clickem sobre dos àtoms i ens apareix la distància entre ells en la finestra del shell)
rasMol> wireframe off (desapareix tota l'estructura; que estigui en format wireframe, que és el que et surt per defecte quan obres un pdb, i el que seleccionis després és el que apareixerà per pantalla)
rasMol> dots <número> (visualitzem una superficie de punts Van der Waals al voltant dels àtoms seleccionats, a una distància regular en una esfera de radi Van der Waals)
rasMol> exit (per sortir del programa)
...i moltes altres, a part de les opcions que ja et surten per
pantalla en forma de menús desplegables
La opció display ens permet diferenciar l’estructura secundària de la molècula amb les opcions strands, ribbons, cartoons,...
Per guardar imatges del rasmol es fa de la següent manera:
Export --> Iris-RGB (és una opció del menú que t’apareix per pantalla)
Et demana que li donis un nom (en la finestra del shell).
La imatge et queda guardada en el mateix directori on es trobava el
pdb.
Per veure la fotografia per pantalla, un cop ja hem sortit de rasmol: $ gimp nom_del_fitxer. Així sobre el programa de tractament gràfic "gimp" i després ja ho guardes amb el format que més et convingui (el que més utilitzem nosaltres és el format JEPG).