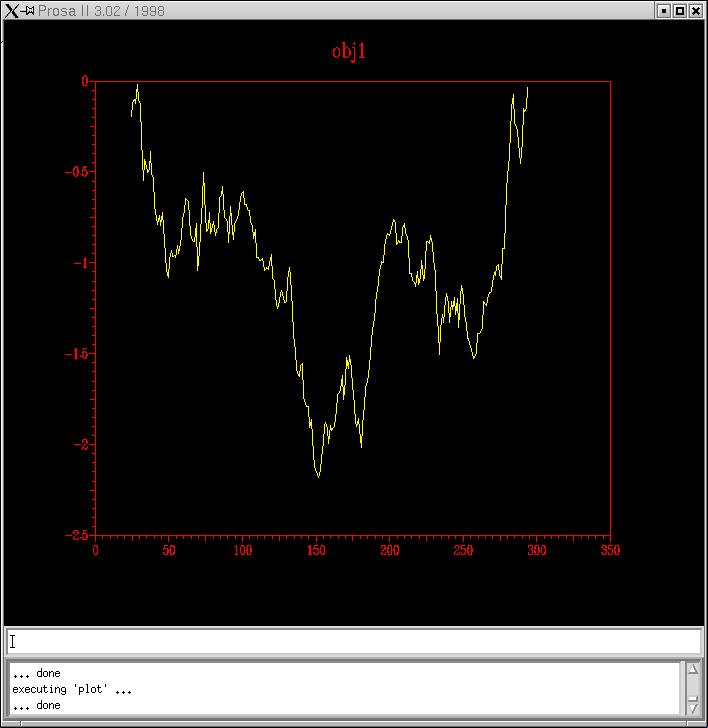
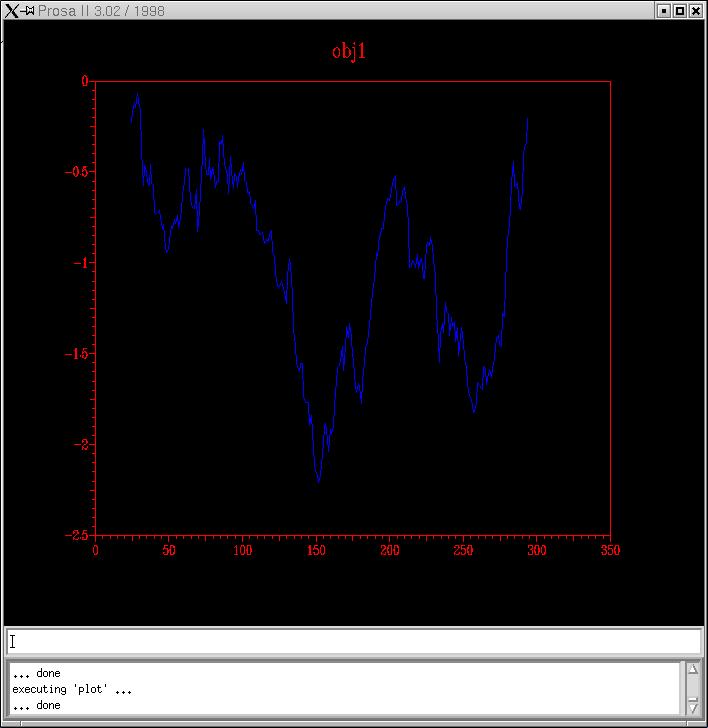
Objectius: validar i identificar regions mal modelades del model de P11018. Per comprovar que una proteïna estigui ben plegada s'utilitza el càlcul de potencials estadístics. En aquesta pràctica aprendrem a utilitzar i interpretar el programa PROSA.
PROSA: És un programa creat per donar suport o ajudar en la determinació del plegament natiu de proteïnes. És útil per determinacions estructurals experimentals i per estudis de modelatge.
La majoria de proteïnes tenen un plegament natiu característic. En condicions fisiològiques aquesta estructura es forma espontàneament. L'estructura plegada de les proteïnes està en funció de la seqüència aminoacídica i del seu entorn natural. En el cas de les proteïnes globulars solubles, l'ambient és generalment una solució aquosa de varis ingredients.
La seqüència d'aminoàcids defineix la identitat molecular d'una proteïna. L'estudi de les normes biològiques, mecanismes moleculars, catàlisis, interaccions moleculars, i moltes més característiques importants de proteïnes concretes, requereixen coneixements sobre les seves estructures tridimensionals.
En general, el càlcul del plegament natiu d'una proteïna a partir de la seqüència aminoacídica és encara impossible. Per això s'han desenvolupat altres mètodes, com el modelatge per homologia, que en molts casos tenen un gran èxit. Però en general, si hom necessita l'estructura nativa de la proteïna no hi ha altra alternativa que l'anàlisis experimental per raig X o l'espectroscopia RMN. Desafortunadament, aquestes tècniques requereix temps i dedicació degut a problemes experimentals (per exemple, per aconseguir cristal·litzar correctament la proteïna en qüestió) i l'únic remei possible és construir un model computacionalment. Evidenment, els resultats dels estudis de modelatge són pitjor que els experimentals, però també és cert que el plegament d'algunes proteïnes cristal·litzades i analitzades per raig X havia resultat ser incorrecte al comparar-ho amb els estudis de modelatge.
Prosa ajuda, primerament, a verificar el model estructural; vindria a ser com un control de qualitat. El programa et calcula uns scores o pels inputs que introdueixes. Les puntuacions dels plegaments de proteïnes natives es troben dins uns límits o rangs d'accpetació. Si l'score que obtinc en passar el meu model pel prosa està fora d'aquest rang, significa que aquella estructura que he predit no és favorable o simplement seria molt difícil trobar-la en la realitat.
Els gràfics d'energies de prosa permeten fer el diagnòstic de zones problemàtiques del plegament. Altes energies corresponen a seccions amb molta tensió estructural (és a dir, amb estrudtura molt forçada i desfavorable). Com més diferències, amb scores i gràfics d'energia, hi hagi entre la forma nativa i el model, més necessitarà aquell model ser millorat.
1. Les comandes bàsiques per començar a evaluar
els quatre models són les següents:
] prosa (ara se'ns obre ja una nova finestra on hi
escriurem directament les comandes i on ens apareixeran cad un
dels gràfics)
] read pdb P11018.B99990001 obj1
] analyse energy obj1 (anàlisis dels potencials
estadístics)
] winsize obj1 50 (creem una finestra de 50 residus per
que en fagi la mitjana dels potencials estadístics i
així aconseguim un gràfic més fàcil
d'interpretar visulament, encara que en realitat estiguem perdent
informació)
] plot
2. Els 4 gràfics d'energies dels models són els
seüents:
P11018.B99990001_ClustalW i P11018.B99990002_Clustalw


Inpertretació: resulta que són molt bons resultats perquè ni tant sols els extrems ens surten amb erebrgies positives, la qual cosa sembla lògica esperar. Són, per tant, models energèticament molt favorables i que tenen moltes probabilitats que un de'ells sigui escollit com a model definitiu. De totes maneres, hi tallarem els extrems igualment, per millorar en el que es pugui. Els dos són molt semblants entre ells. Podríem veure una petita diferència en el 02 (color blau), on sembla que els pics són lleugerament més alts, menys negatius. Però no deixa de ser irrellevant.
P11018.B99990001_HMM i P11018.B99990002_HMM
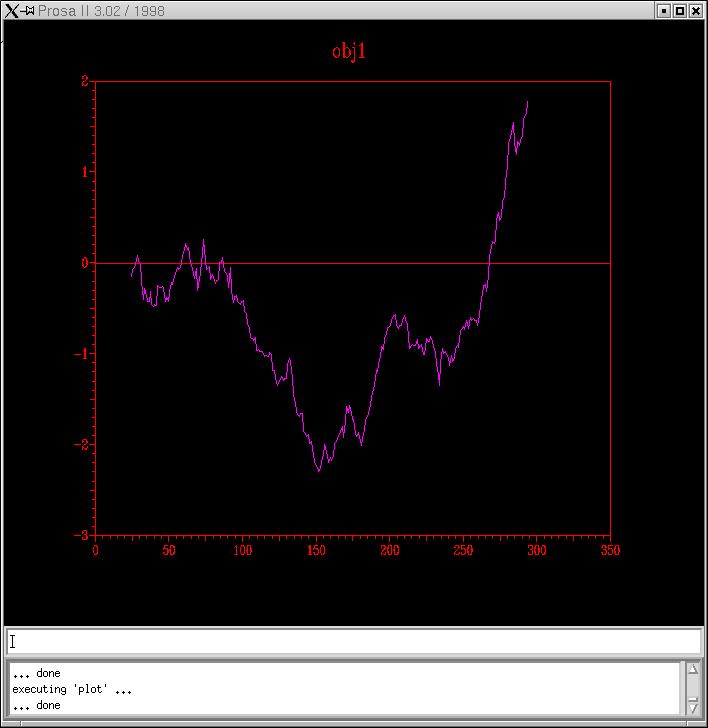
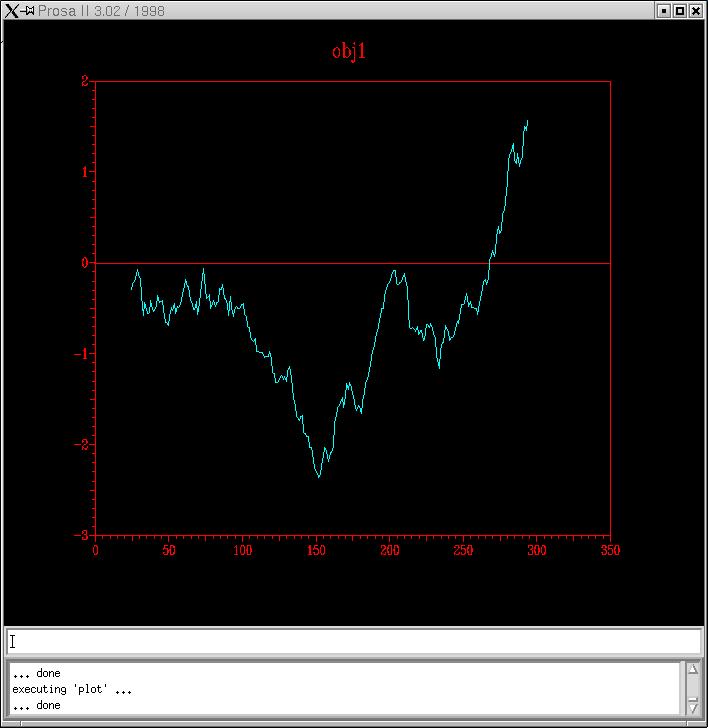
InterpretacióEsperaríem més aviat uns resultats com els de HMM, én a dir, amb energies positives als extrems. De fet, els gràfics dels resultats del Clustal sós sorprenenment molt bons. De totes maneres, tallarem els extrems per als quatre models. Per què és normal trobar extrems amb energies positives? És degut a que no hem trobat templates que cobrissin aquestes dues zones i per tant, al fer-se el modelatge, no s'hi ha pogut predir estructura secundària i aquesta organització dels extrems é,s molt desfavorable.
3. Per tant, tallem els extrems de la proteïna P11018.
Eliminem tots els residus que no estiguin alineamts amb cap
template. En el meu cas he tret 9 residus de l'extrem N terminal
i 6 del C. El següent pas és tornar a analitzar-ho
amb prosa per veure si hem eliminat el problema o si
contràriament, haurem de fer més retocs. Els
gràfics que esperem trobar són d'energies
més negatives.
P11018.B99990001_Clustalw i p11018.B99990002_Clustalw
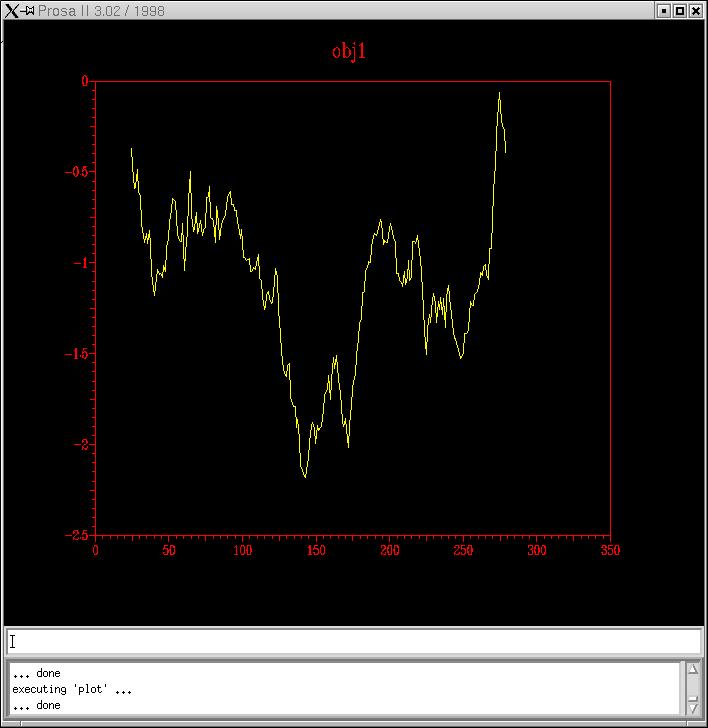
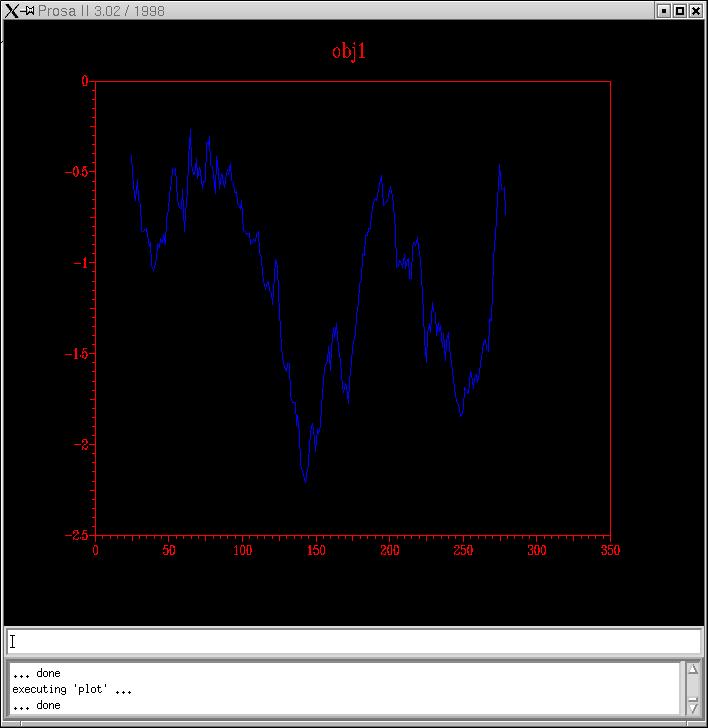
p11018.B99990001_HMM i p11018.B99990002_HMM
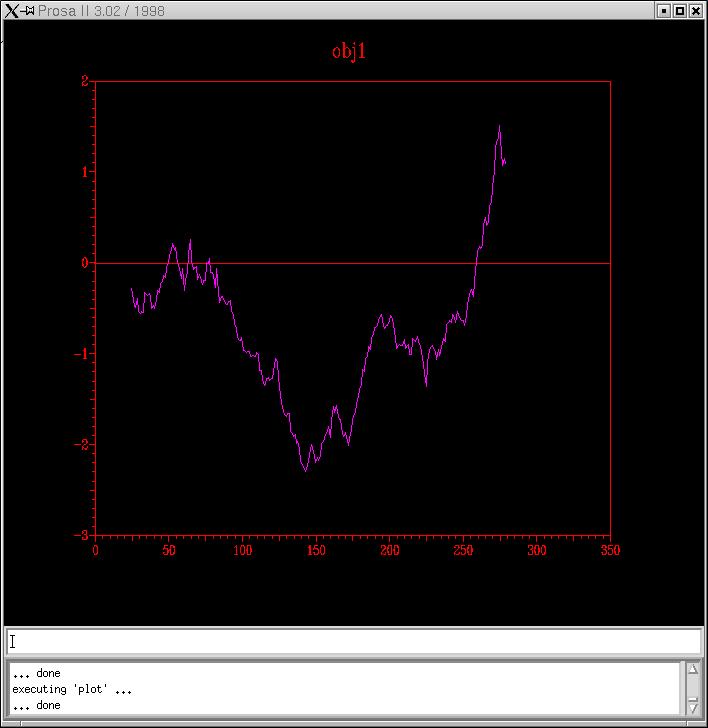
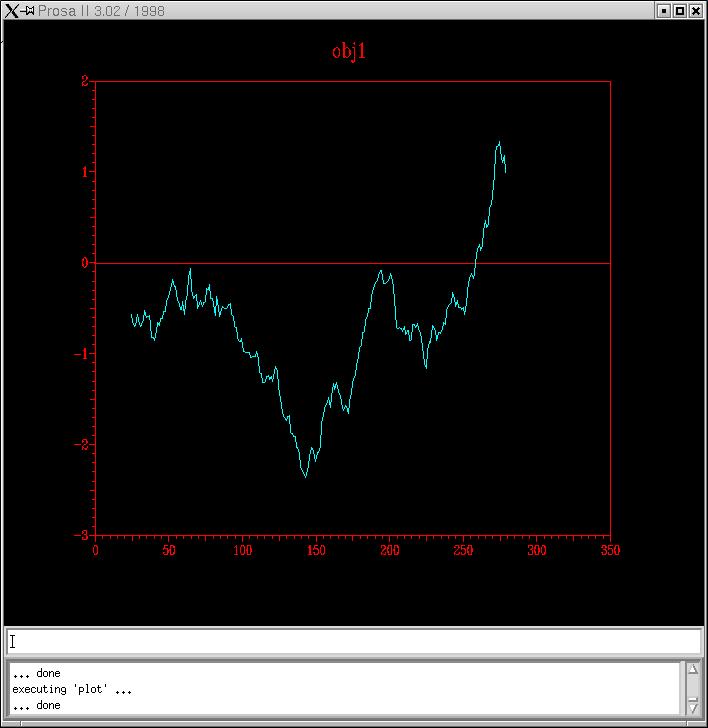
Conclusion: Efectivament, els models han millorat. Observant els gràfics podem fer una primera elecció de model descartant-ne dos: els de Hidden Markov. I dels altres dos, resusltats de Clustal, podem veure que el model 02 és lleugerament millor que le 01. Amb la següent pràctica acabarem d'escollir el definitiu.