Pràctica
5.4- Modelat de seqüències IV: Evaluació del model amb Procheck
Pràctiques de Biologia Estructural-
Carlos Masdeu Ávila- Nia=16965
<< >>
Ara que ja tenim models creats, utilitzarem el software Procheck per evaluar
si els angles i forces de torsió dels àtoms són possibles,
a més de si hi ha superposicions espacials, etc...
Comprovació de la mínima resolució
Per utilitzar-lo,
hem de mirar abans quina és la resolució més grollera d'entre
totes les estructures que han contribuit a formar el model.
En el nostre cas, la resolució corresponent a cadascuna de les estructures
és:
'1ak9' 1.8
'1scjA' 2.0
'1ubnA' 2.4
'1sca' 2.0
(Per tant, la més
grollera és 2.4)
Execució
de Procheck
Procheck es pot cridar de varies maneres. Es pot crear un arxiu 'list' on constint
els noms de les estructures a analitzar, i també es pot cridar de la
següent manera (per analitzar només una estructura):
procheck_single <estructura>
resolució
Per avaluar cada model,
procheck emet 10 arxius en format postscript i un resum en format text (amb
el format *.sum).
Outputs
de Procheck
Com s'ha mencionat prèviament,
Procheck emet 10 outputs en format postscript:
1. Ramachandran plot
2. Gly & Pro Ramachandran plots
3. Chi1-Chi2 plots
4. Main-chain parameters
5. Side-chain parameters
6. Residue properties
7. Main-chain bond length distributions
8. Main-chain bond angle distributions
9. RMS distances from planarity
10. Distorted geometry plots
Tots
són informatius d'un paràmetre o altre important per avaluar correctament
el model, però aquí només analitzarem dos: Ramachandran
y el resum (summary)
Representació
en plot de Ramachandran:
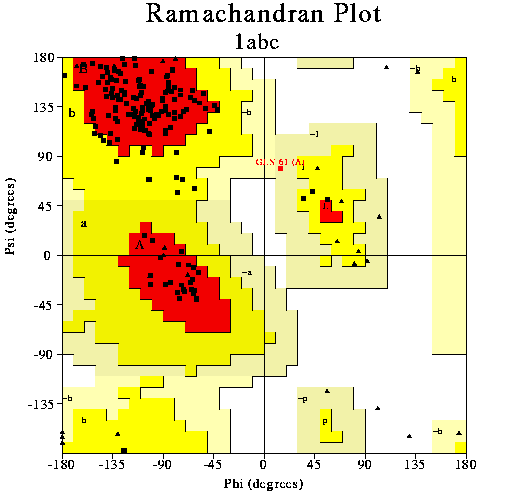 |
Aquesta
representació situa, sobre l'eix Y, els graus de l'angle Psi, i
a l'eix X, els graus de l'angle Phi de l'enllaç peptídic
per a cada residu de la proteïna. Així, podem situar sobre
les dos dimensions qualsevol residu, en funció del grau dels dos
angles abans mencionats.
La utilitat d'aquesta representació
resideix en què està descrit quines posicions són
estèricament 'permeses' i quines no, de tal manera que en funció
del tipus de zona on s'ubiqui el residu que estem avaluant, podem predir
si està ben model·lat o no.
Una proteína perfectament
model·lada no deuria tenir gaire cap ocurrència a les zones
'blanques' ('NOT ALLOWED') i molt poques a les zones beix, tal com mostra
la foto d'exemple.
Quan un residu està en
una posició no permesa o 'incòmoda', Procheck ens el pinta
de vermell i ens diu tipus de residu i nombre, perquè el podem
localitzar i corregir (si es pot).
|
| |
|
Summary:
+----------<<< P R O C H E C K S U M M A R Y >>>----------+
| |
| clustal2.mod 2.4 319 residues |
| |
*| Ramachandran plot: 88.2% core 10.0% allow 0.7% gener 1.1% disall |
| |
*| All Ramachandrans: 10 labelled residues (out of 317) |
+| Chi1-chi2 plots: 4 labelled residues (out of 180) |
| Main-chain params: 6 better 0 inside 0 worse |
| Side-chain params: 5 better 0 inside 0 worse |
| |
*| Residue properties: Max.deviation: 4.6 Bad contacts: 9 |
*| Bond len/angle: 6.5 Morris et al class: 1 1 2 |
+| 1 cis-peptides |
| G-factors Dihedrals: -0.05 Covalent: -0.26 Overall: -0.12 |
| |
| M/c bond lengths: 98.8% within limits 1.2% highlighted |
*| M/c bond angles: 92.6% within limits 7.4% highlighted 2 off graph |
| Planar groups: 100.0% within limits 0.0% highlighted |
| |
+----------------------------------------------------------------------------+
+ May be worth investigating further. * Worth investigating further.
|
El summary es un arxiu en format *.sum
que ens resumeix les dades de tots els paràmetres que han estat calculats,
i ens ho presenta de manera breu perquè es pugui fer una avaluació
ràpida.
D'entre tots els valors que apareixen, els més importants són:
*Ramachandran
plot: (corresponent a la representació continguda als arxius
*_01.ps). Indica els percentatges de residus que es troben a cadascuna de les
regions:
core (residus que estan en posicions
favorables)
allow (residus que estan en posicions
permeses)
gener (residus que estan en posicions
permeses, si som "generosos")
disall (residus que de cap manera
estan en posicions permeses)
*Bad contacts: Ens indica quants àtoms
estan en posicions en les que col·lisionarien amb un altre
