

En aquesta pràctica realitzaré alineaments estructurals mitjançant el programa STAMP, que fa l'alineament estructural sense alineament de seqüència previ.
Alineament de Globines
Altres alineaments: dominis β-propeller
Per fer l'alineament de les globines primerament crearé l'arxiu .domains, que STAMP precisa per executar l'alienament. Aquest arxiu conté el nom dels pdb i la cadena que s'ha d'usar (veure arxiu.domains).
A continuació executaré Stamp:
stamp -l globin.domains -rough -n 2 -prefix globin Amb això, Stamp crea 5 arxius globin.1 a globin.5 que contenen els progressius alineaments. Per visualitzar l'alineament, prendré l'arxiu globin.5 (veure arxiu), ja que aquest conté l'alineament definitiu (els altres contenen els passos intermitjos). Així doncs, transformaré aquest arxiu per fer-lo visualitzable amb RasMol:
transform -f globin.5 -g -o globin.5.pdb Ara, l'alineament és visualitzable amb RasMol.

click to enlarge A partir del fitxer globin.5 també podem obtenir un alineament de seqüència en format Clustalw (veure alineament) mitjançant el programa aconvertMod2:
aconvertMod2.pl -in b -out -c globin.5.clw
Altres alineaments: dominis β-propeller
A continuació realitzaré un alineament amb STAMP de proteïnes amb dominis β-propeller. Primerament, cercaré 4 proteïnes a SCOP per fer l'alineament:
DOMINIS β-PROPELLER SELECCIONATS PER L'ALINEAMENT Classe Tot β Plegament 7 bladded β-propeller Superfamília Quinoprotein amino dehydrogenase, catalytic subunit Família Methylamine dehydrogenase H chain Estructures 2mta chain H - Paracoccus denitrificans
Família Quinohemoprotein amino dehydrogenase B chain Estructures 1jju chain B - Paracoccus denitrificans
Superfamília Trp-Asp repeat Família Trp-Asp repeat Estructures 1b9y chain A - Bos taurus
1gxr chain B - Homo sapiens
Una vegada escollides les estructures crearé l'arxiu .domains (veure arxiu.domains), i executaré Stamp:
stamp -l betaprop.domains -rough -n 2 -prefix betaprop Finalment, obtindré l'alineament en format Clustalw (veure alineament) i el fitxer visualitzable amb RasMol:
transform -f betaprop.3 -g -o betaprop.3.pdb aconvertMod2.pl -in b -out -c betaprop.3.aln
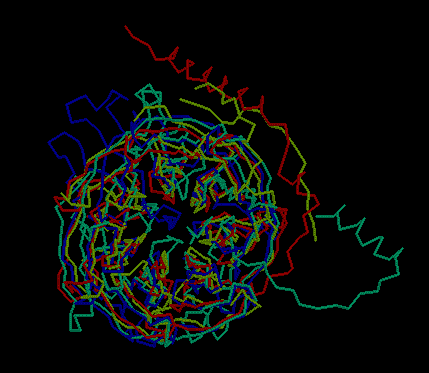
click to enlarge En aquest cas, l'alineament és molt dolent, segurament degut a la diferent longitud de les seqüències.

