LŽobjectiu de la pràctica ha estat el fet de familiaritzar-nos amb el paquet de programes del Silicon Graphics Station. Els programes utilitzats durant la pràctica, han estat bàsicament dos: Rasmol i GIMP
Rasmol és un programa dissenyat per a visualitzar estructures en format .pdb (entre dŽaltres possibles formats) i poder-les modificar per tal de destacar-ne els detalls més rellevants: estructura, cadenes, tipus de plegament...
GIMP és un programa que hem utilitzat per captar les imatges tractades utilitzant Rasmol, i poder-les importar en altres formats (.gif, .jpg ...) per tal de poder-les utilitzar com a imatges en altres documents o pàgines web.
Les comandes que hem utilitzat durant la pràctica han estat les següents:
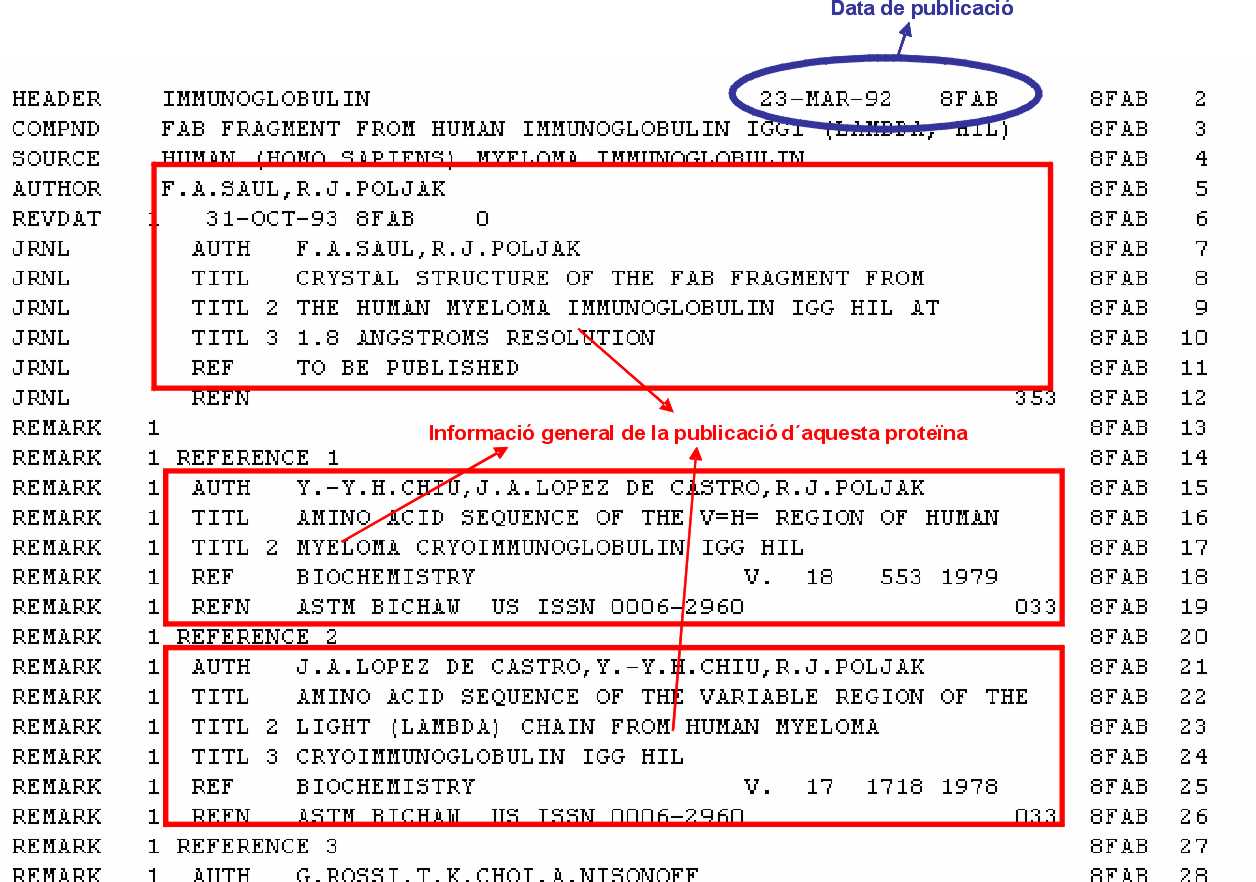
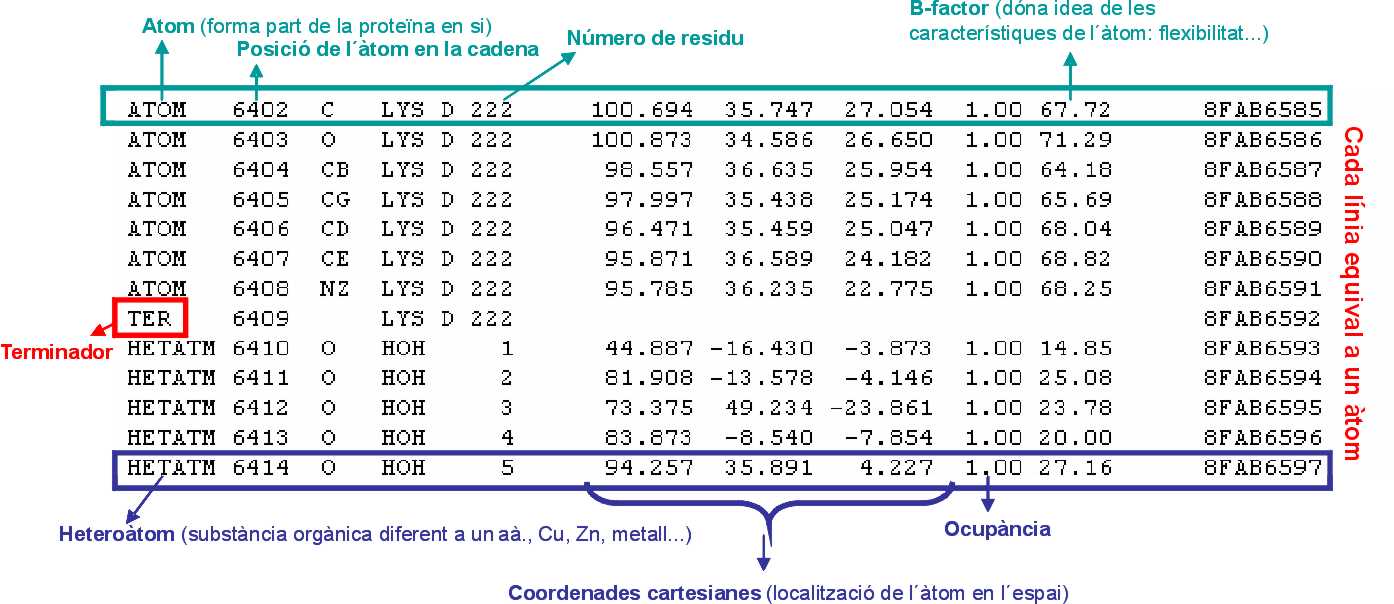
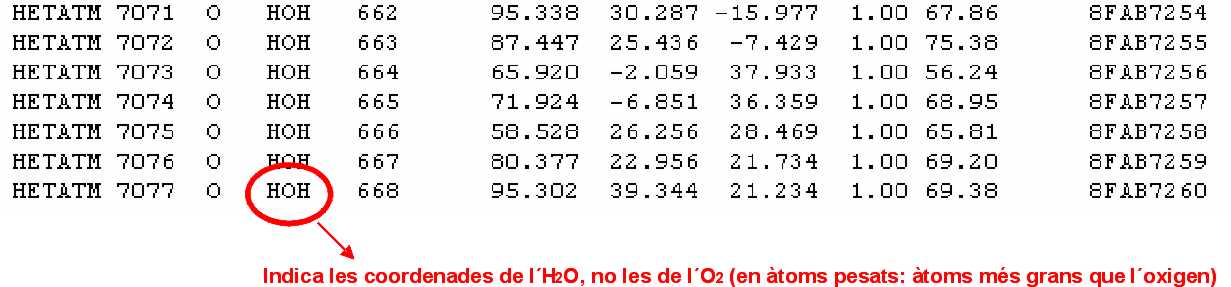
Ara estudiarem la informació que conté un arxiu en format .pdb. Un arxiu en aquest format, mostra molta informació:
En aquests documents, també hi podem trobar informació sobre la proteïna que estem estudiant:
O també:
Ara visualitzarem les proteïnes escollides amb el programa Rasmol i les anirem modificant per tal dŽanar familiaritzant-nos amb el programa.
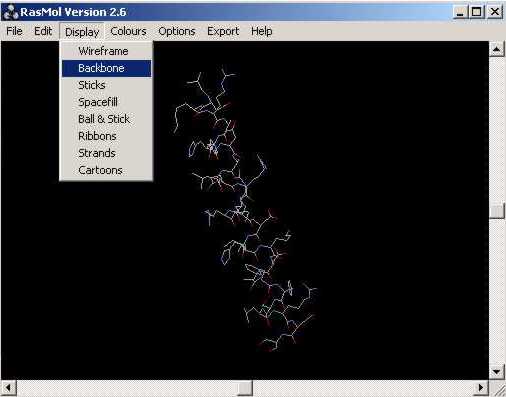
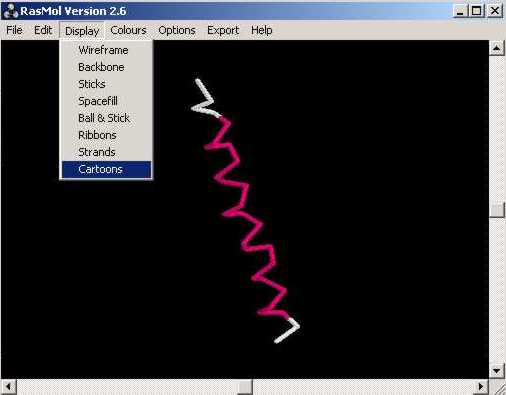
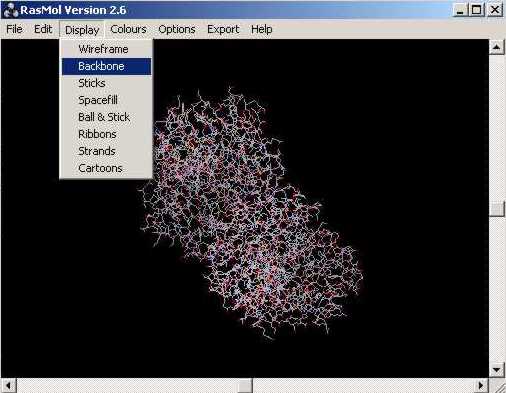
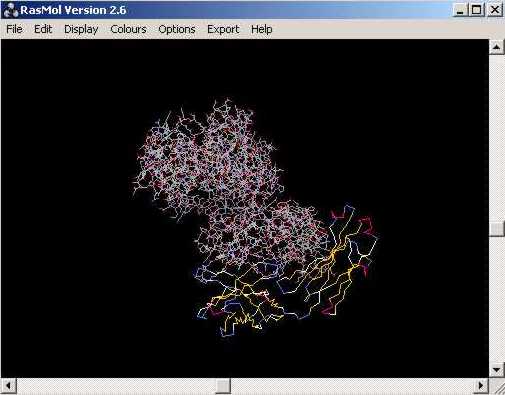
En obrir un arxiu .pdb o .ent amb lŽaplicació, es mostra (p.e. pdb1an2.ent):
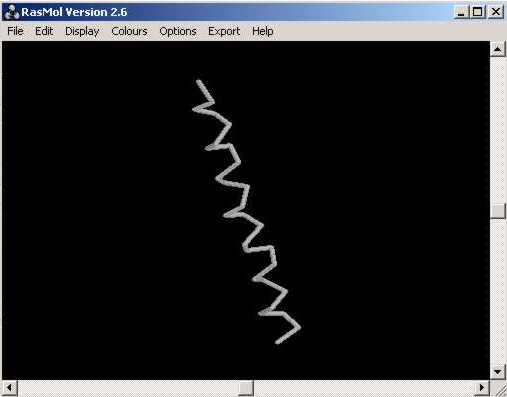
A partir dŽaquí i seguint els passos que a continuació sŽindiquen, podem arribar a retocar la imatge:
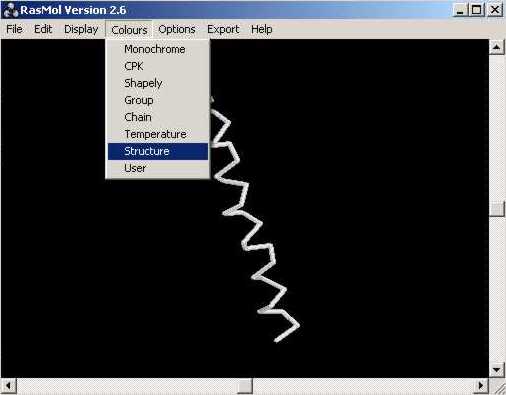
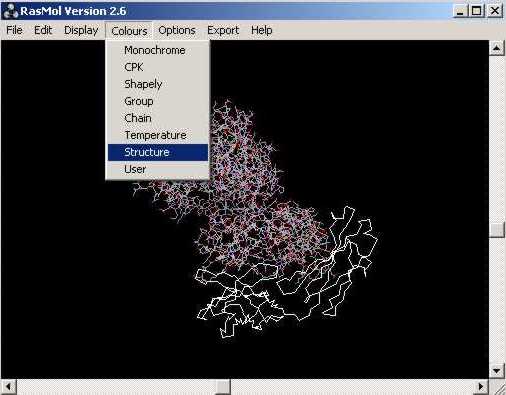
També podem modificar el color de lŽestructura...:
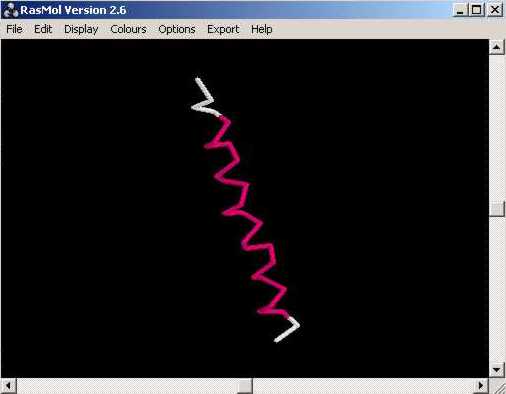
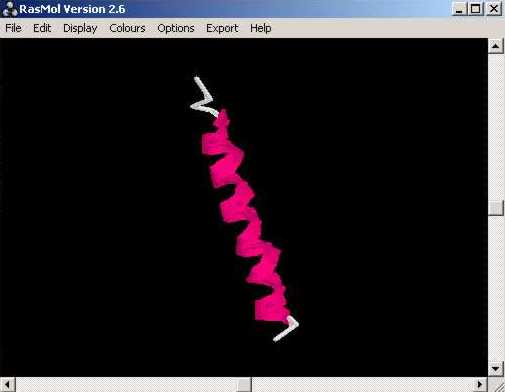
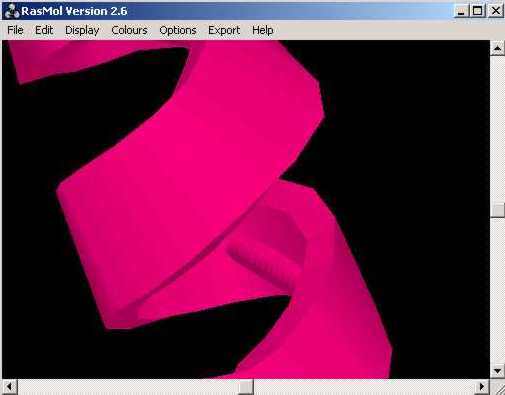
... així com mostrar el tipus de plegament de la proteïna en qüestió:
Amb aquest programa de visualització, podem modificar lŽestructura de tal forma que es faci petita o gran per a detallar certs aà o certs possibles llavis dŽactivació, etc. així com rotar lŽestructura.
Per a poder allunyar o apropar una estructura, apretem alhora el botó shift i el botó esquerre del mouse tot tirant amunt o abaix per allunyar o apropar lŽestructura, respectivament.
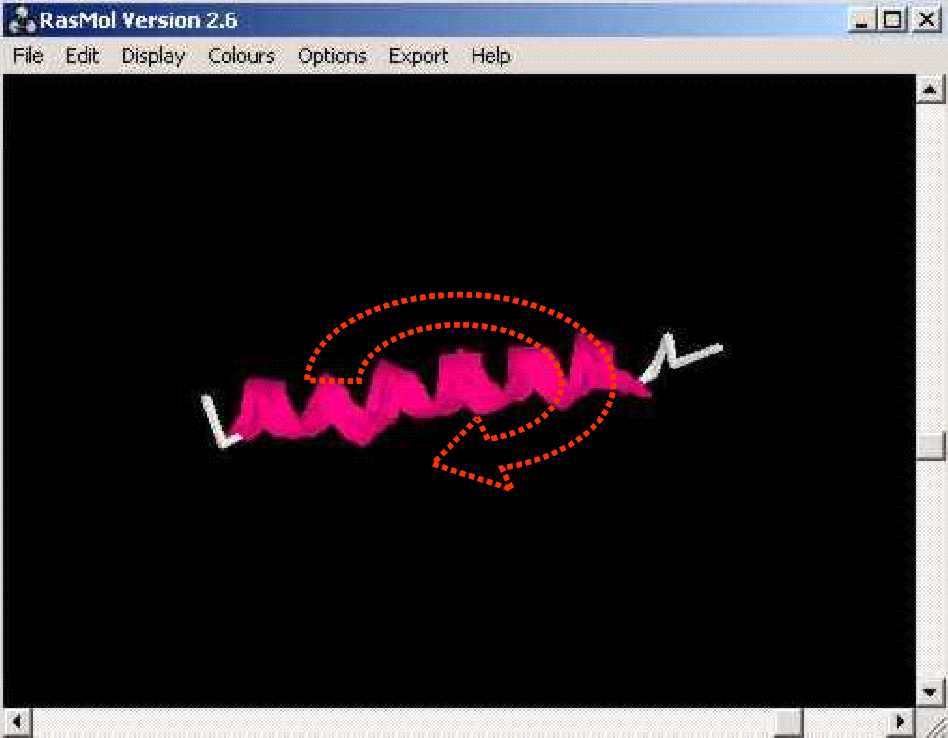
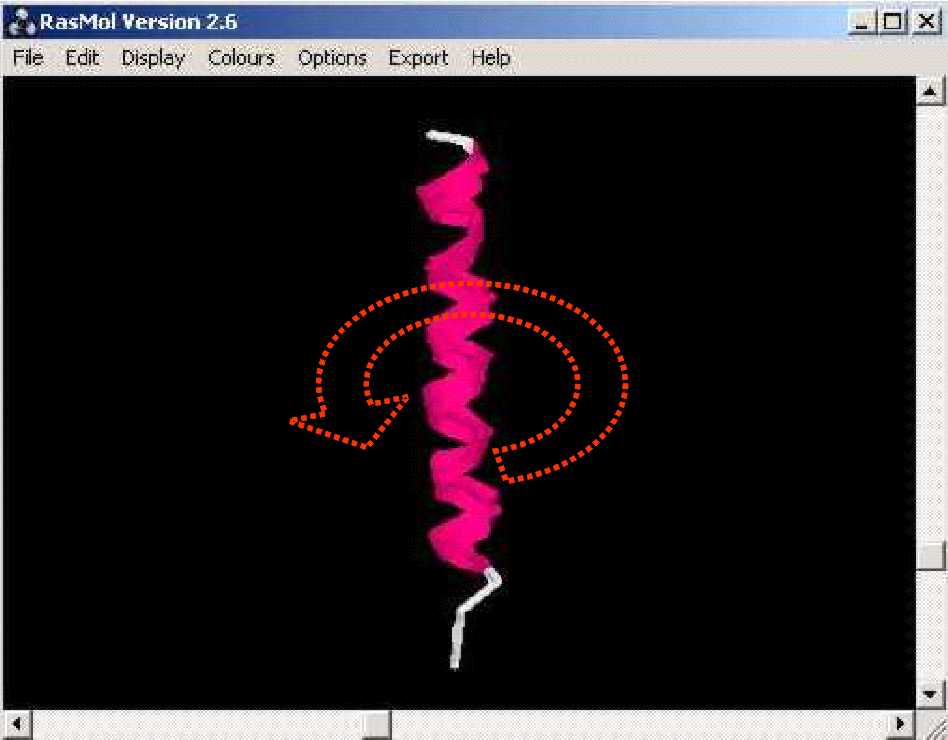
Per girar cap a lŽesquerre o cap a la dreta una estructura, mourem lŽscroll que hi ha al marge dret de la finestra del programa.
...apropem... ...cap a la dreta... ...cap a lŽesquerre...
Ara agafem una altre arxiu per tal de revisar més comandaments(p.e.pdb8fab.ent):
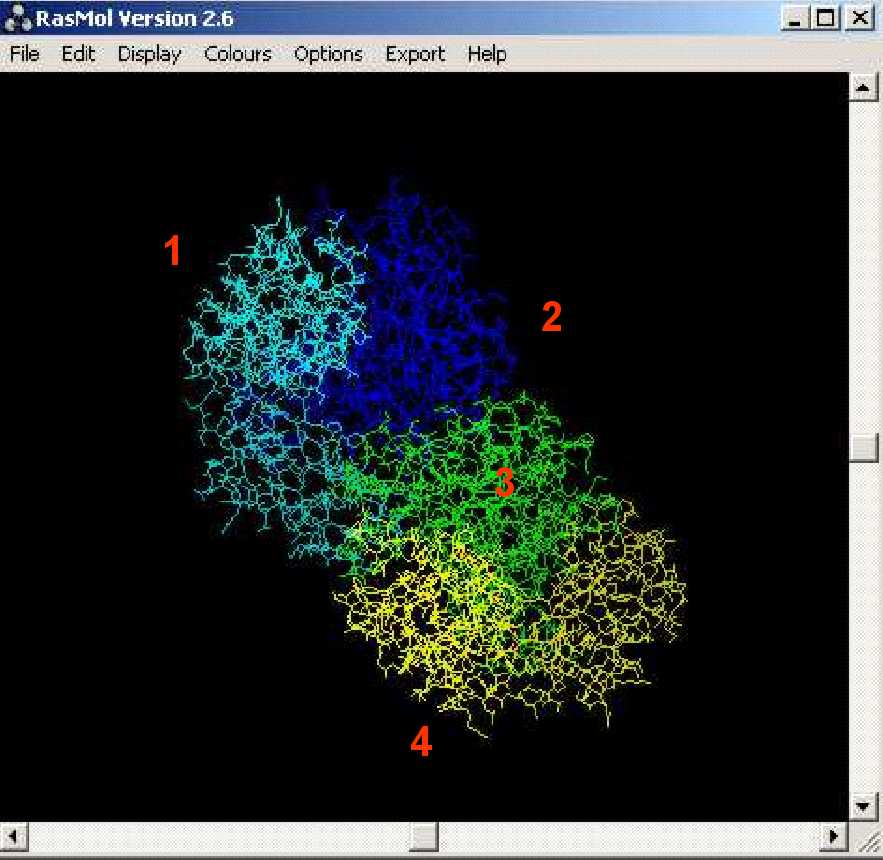
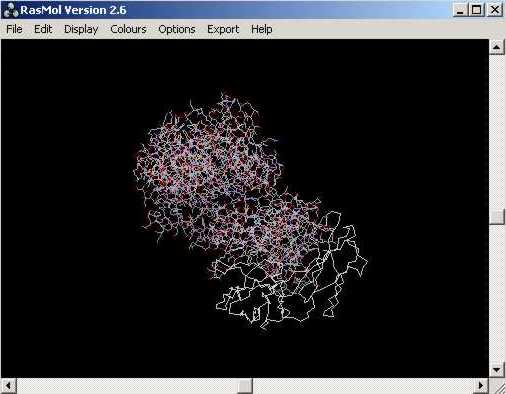
Comprovem que aquesta proteïna presenta quatre cadenes...
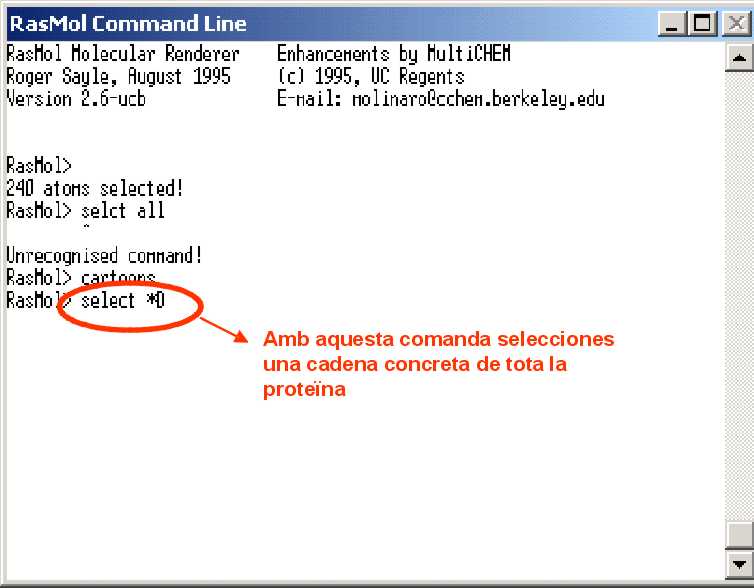
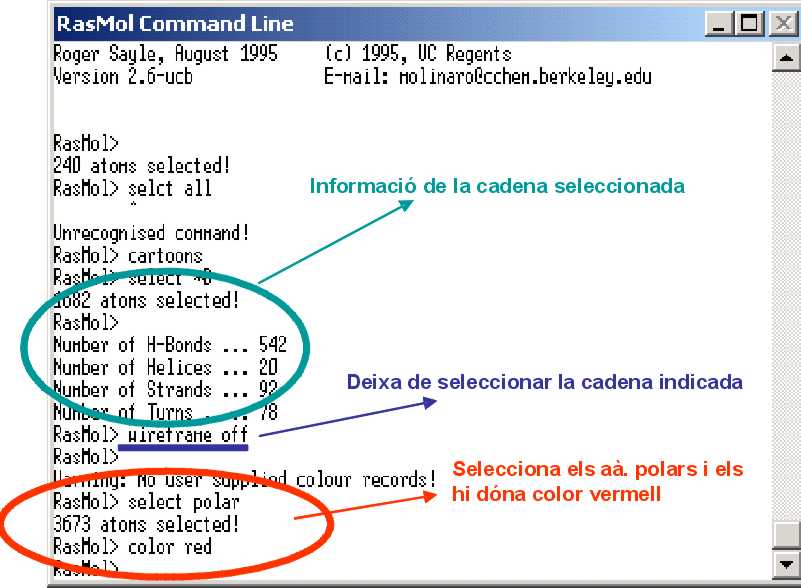
Ara es tracta de seleccionar una sola dŽaquestes quatre cadenes i...:
...donar-li format i...:
...també color:
Per a deixar de seleccionar una cadena determinada usarem la comanda wireframe off:
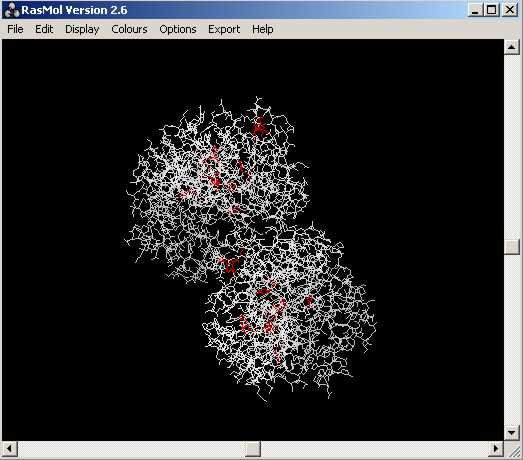
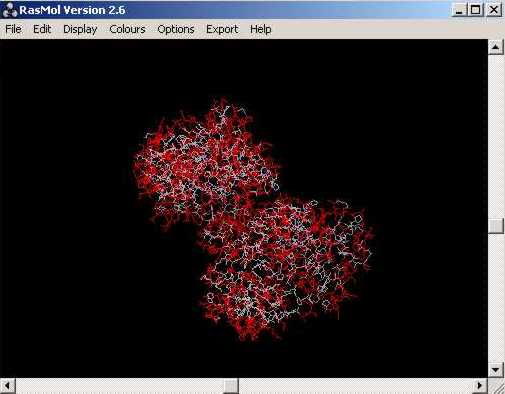
Amb aquesta última comanda aconseguim seleccionar només ela aà. polars i a més destacar-los de la resta, tot aplicant-los-hi color vermell.
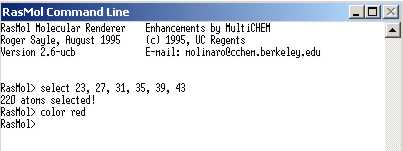
Hi han dŽaltres comandes que permeten seleccionar un sol aà i a més donar-li un determinat format per a localitzar-lo dins la proteïna:
Primer hem seleccionat una sola cadena dŽentre les altres quatre. A continuació li hem donat color groc i hem procedit a seleccionar-ne un sol residu que destacarem a continuació amb color blanc. Els següents pasos a seguir, per tal dŽobtenir la imatge, han estat: hem seleccionat en el menú Display lŽopció Sticks i a continuació del menú Options, Labels.
...i el resultat ha estat:
$ cp -r /disc9/practica_1/PDB/pdb1an2.ent .
$ cp -r /disc9/practica_1 .
Per a escriure un fitxer hem dŽutilitzar un editor de text (emacs, kwrite, vi...) per tal dŽintroduir-hi el contingut que desitgem. Per a crear un arxiu es pot cridar lŽeditor de text, escriureŽn el contingut i guardar-lo donant-li un nom, o bé, podem cridar lŽeditor de text donant-li un nom, per exemple: $ emacs prova.pl $ kwrite prova.fa ... LŽextensió (*.*) que li donarem a lŽarxiu que estem creant dependrà de la informació que hi estem introduint: un programa, seqüències aliniades...
Per a canviar el nom dŽun fitxer donat (nomfitxer1) per un altre nom (nomfitxer2), cal fer: $ mv nomfitxer1 nomfitxer2 Per a canviar el nom dŽun directori (dir1) per un altre nom (dir2), cal fer: $ mv dir1 dir2
Per a canviar de lloc un arxiu o directori: $ mv fitxer /root $ mv dir /root Només cal indicar lŽarxiu o directori que volem desplaçar i a continuació, la ruta del directori on hi afegirem aquest arxiu o directori.
Les variables dŽentorn, tal com el seu nom indica, són variables que cada usuari pot definir (per tant poden ser diferents entre els diferents usuaris), i que permeten personalitzar lŽentorn de LINUX a gust de lŽusuari. Les variables dŽentorn ens faciliten la feina ja que ens permeten estalviar comandes llargues que es repeteixen sovint. $ alias nom='(ruta del directori al que es preten accedir)' $ nom Per exemple: $ alias rasmol= '/disc9/bin/rasmol/' $ rasmol ...i amb això, sŽexecuta el programa Rasmol. Altres variables dŽentorn que hem utilitzat (a part de les definides en Bash pel propi usuari), han estat les predefinides en tcsh dins de disc9 (source /disc9/cshrc).
Per a exemplificar algunes comandes del Rasmol, utilitzaré lŽarxiu pdb8fab.ent abans utilitzat per a mostrar algunes comandes del programa.
Per a seleccionar una determinada regió o bé aà. sols, els hem de seleccionar (select i seguit el número dŽàtom determinat o lŽinterval de la regió a seleccionar) i a continuació els hi afegim un color per a resaltar-los de la resta.
Opció força útil per a calcular la distància entre dos residus (per exemple, per a determinar la possible formació dŽun pont S-S és útil saber la distància a la que es troben els dos àtoms de sofre de les dues Cisteïnes allunyades). LŽopció que hem dŽutilitzar per a calcular la distància existent entre dos residus, és: pick distance i tot seguit assenyalar els residus que volem determinar. A continuació la pantalla et mostra la distància en Å, a la què es troben.
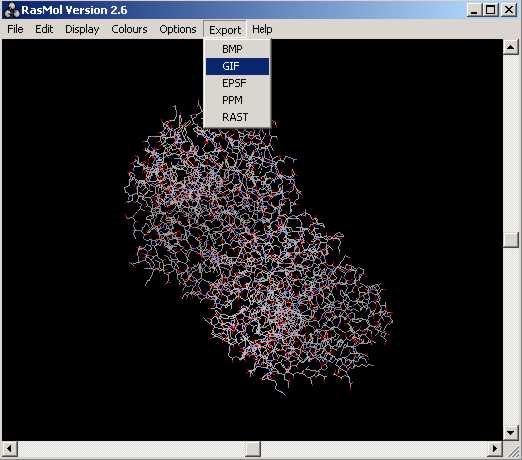
Per a guardar imatges només cal exportar la imatge a un format .gif i desar-la al disc o bé importar aquesta estructura a un programa de tractament de imatges (GIMP...), per tal de modificar-la...
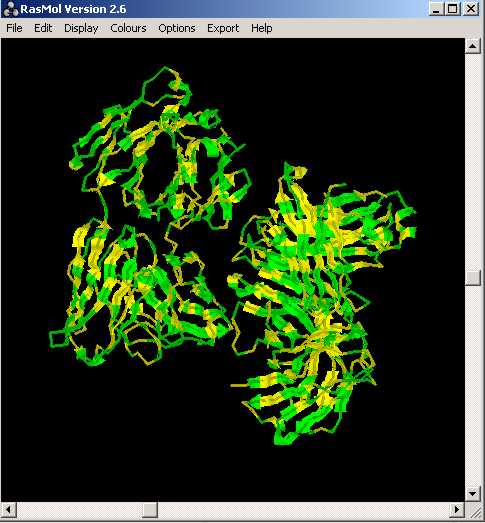
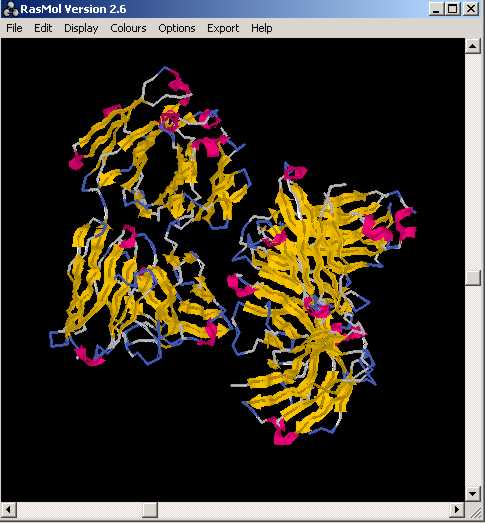
Seleccionem aà.polars amb la comanda select polar, els hi donem color verd (color green). A continuació seleccionem els aà. hidrofòbics (no polars) amb la comanda select hydrophobic i els hi donem color groc (color yellow) i ara ho contrastem amb lŽestructra colorejada amb color strucutre: DŽentre els residus polars marcats hi podem trobar els següents aà.: Arg, Asn, Asp, Cys, Glu, Gln, His, Lys, Ser, Thr. Mentre que dŽentre els residus hidrofòbics, hi poden ser: Ala, Gly, Ile, Leu, Met, Phe, Pro, Trp, Tyr, Val.
$ cp -r /disc9/practica_1 .
$ cd practica_1
$ ls
PDB SELECT
$ cd PDB
$ emacs pdb1an2.ent (veure el contingut del fitxer)
$ emacs pdb8fab.ent (veure el contingut del fitxer)
$ alias rasmol='/disc9/bin/rasmol'
$ rasmol pdb1an2.ent & (més endavant veurem lŽestructura)
$ rasmol pdb8fab.ent & (més endavant veurem lŽestructura)

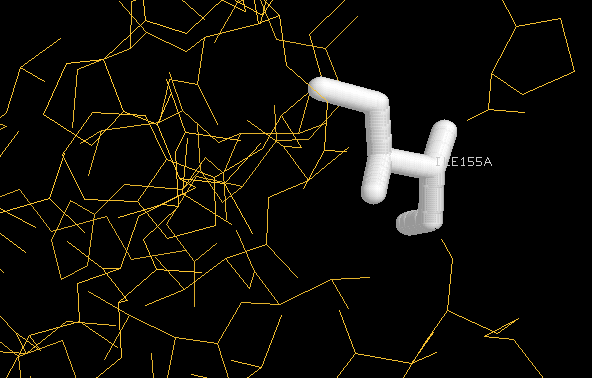
(Copia lŽarxiu pdb1an2.ent que es troba a la carpeta practica_1 dins la subcarpeta PDB al directori actual on ens trobem)
(Copia el directori practica_1 al directori actual on ens trobem)