




|
Prosa, és un programa útil que ens pot ajudar a establir una qualitat de control per a l'estructura de la proteïna que estem estudiant així com per a estudis de modelatge proteic.. Els gràfics d'energia produïts per Prosa no mostren l'energia total del sistema, sinó que ens proporcionen dades de subenergia de les diferents regions de l'estructura (energia entre parells d'aà, energia de superfície i energia combinada) Els gràfics d'energia que genera el programa, proporcionen una eina de diagnòstic per aquelles regions problemàtiques de l'estructura en qüestió: les zones on l'energia és elevada (valor positiu) corresponen a regions sotmeses a estrés o regions amb problemes en el seu plegament; mentre que a mesura que els gràfics d'energia s'acosten a valors enegètics similars als de la proteïna nativa, els fa més favorables i per tant l'estructura serà millor.
En el transcurs de la pràctica, hem emprat les següents comandes:
 ... i en el display del programa, anem introduint les comandes: Primer carregarem un arxiu .pdb per tal de calcular-ne les subenergies:
A continuació canviarem el tamany de la finestra (en aquest cas el tamany de la finestra de 50 residus):
I ara per tal de d'editar el gràfic:
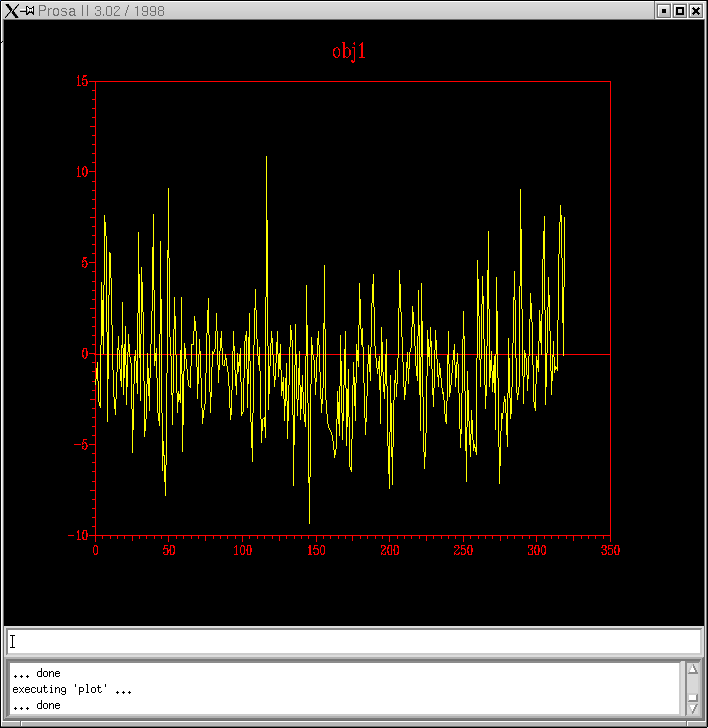
... i amb això es mostren les diferents energies: pair; surface i combined...
Un cop introduïdes les comandes bàsiques per a carregar qualsevol .pdb i un cop visualitzades les diferents subenergies de qualsevol estructura, ens podem centrar ja en els nostres models en concret: ...el pas següent és determinar els colors de les diferents energies per a cada una de les estructures: Per als models probl1amb3.ent i probl1amb3_2.ent:
... i per a les estructures probl2amb2.ent i probl2amb2_2.ent:
Altres comandes també utilitzades al llarg de la pràctica:
... per dibuixar totes les energies de les proteïnes carregades:
...per a sortir:
De la present pràctica se'n dedueix:
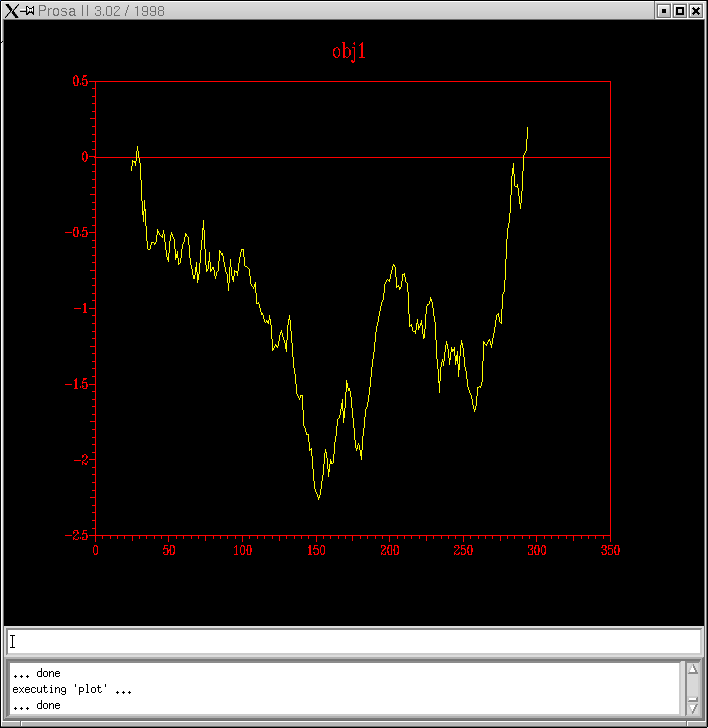
Aquestes imatges mostren les diferents pair energy (energia que apareix per defecte) de cadascun dels models obtinguts tant per a la pràctica 5.1 (Clustalw), com per al model de la pràctica 5.2 (HMM). ... en groc es destaca lŽenergia per al submodel probl1amb3.ent i en vermell, el submodel probl1amb3_2.ent:
... de forma similar a la imatge anterior: en groc destaco lŽenergia per al submodel probl2amb2.ent i en vermell, el submodel probl2amb2_2.ent:
Vistes les dues anteriors il·lustracions puc concloure quin dels dos submodels per a cada model és millor; així doncs en el cas del primer model (pràctica 5.1: Clustalw), el millor és el probl1amb3_2.ent i en el cas del segon model (pràctica 5.2: HMM), el probl2amb2.ent.
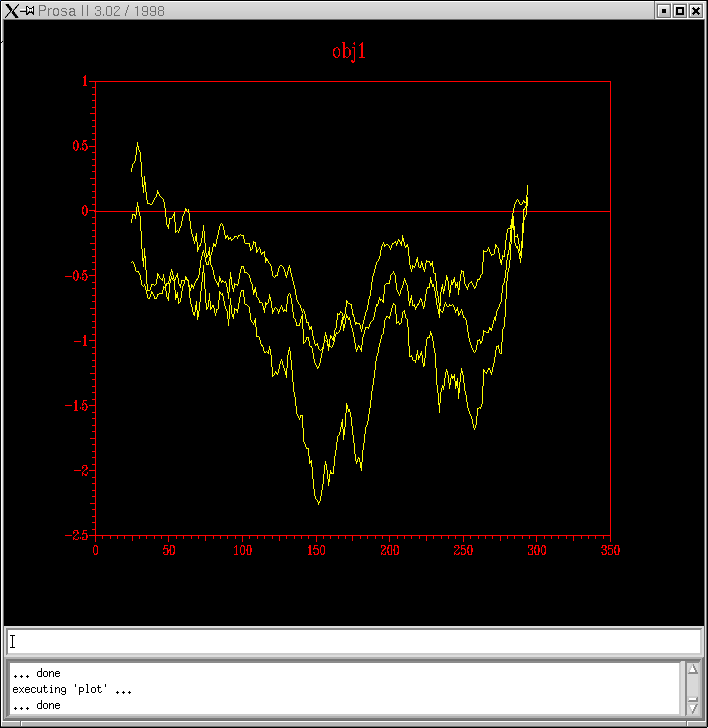
... i finalment, en verd es mostra el submodel probl1amb3_2.ent i en blau, el submodel probl2amb2.ent.
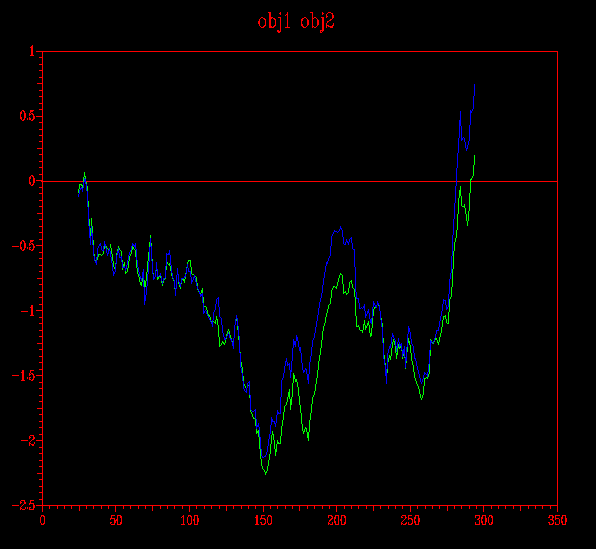
DŽaquestes imatges nŽextrec la conclusió que el model obtingut a partir del Clustalw (pràctica 5.1) és millor que l'obtingut per a HMM (pràctica 5.2) i que dŽaquest mateix model, el millor dels dos submodels és el probl1amb3_2.ent.
PSI-Pred, és un programa que ens facilita un mètode de predicció de l'estructura secundària, tot basant-se en xarxes neuronals (no les hem vist encara a teoria), les quals realitzen un anàlisi dels resultats obtinguts a partir de PSI-Blast (Position Specific Iterated - BLAST).
Per tal d'arribar a calcular l'energia global del sitema no és vàlid el programa anterior, ProsaII, i és en aquest punt quan cal introduir les comandes emprades en l'execució de PSI-Pred.
Partint d'un model (estructura .pdb), hem de seguir les següents instruccions (veure pràctica 5.4):
La comanda dssp, prediu l'estructura secundària que hauria de presentar el model de forma teòrica:
... i els resultats que n'obtenim per a cada un dels models:
D'aquests arxius se'n pot observar l'estructura que adopten els diferents residus al llarg de la seqüència (T i S per a girs o loops, H per a les hèlixs α i E per a les làmines β...).
Amb aquest programa (aliss.pl), aconseguim transformar un arxiu .dssp a un arxiu .pir. Aconseguim passar d'un arxiu en format vertical (.dssp) a una arxiu en format horitzontal (.pir).
Un cop més els arxius de sortida:
I finalment amb aquest altre programa, aconseguim passar d'un arxiu .pir a un arxiu .clw (aliniament).
Els arxius de sortida:
... però en canvi si partim d'una sola seqüència (per exemple partim una seqüència de la qual no en coneixem la seva estructura secundària perquè no està cristal·litzada com pot ser el cas de la seqüència de la subtilisin, P11018.fa):
De l'execució d'aquest programa, surten diversos arxius de sortida: .ss2, .horiz,...Els arxius .ss2, contenen informació: C de random coil (tot allò que no té estructura secundària), E de làmina β i H d'hèlix α
La següent comanda passa un arxiu .ss2 a un arxiu .pir, de manera similar que el programa anterior aliss, converteix un arxiu amb format vertical a un arxiu en format horitzontal.
L'execució del programa PSI-Pred, partint d'una seqüència en format fasta, dóna com a resultat un arxiu que conté la seqüència i una predicció (apareixen C, H i E). En canvi, a l'executar la comanda dssp a partir d'una estructura (model), n'obtinc una seqüència i un càlcul (arxiu .pir). Els arxius, en aquest cas, presenten una altra anotació per a les estructures:S, T, B... (veure diagrama explicatiu) El que realment ens interessa és aconseguir un arxiu que contingui: la seqüència, la predicció de l'estructura secundària i el càlcul de l'estructura secundària per a cada un dels models, en un sol arxiu i sense informació repetida. Podem obtenir aquest arxiu tot editant cada arxiu .pir, esborrant el que es repeteix i després enganxant-lo en un únic fitxer o bé podem juntar tots els arxius .pir a través de la comanda cat, i després esborrar-ne la informació que es repeteixi. Un cop haguem obtingut aquest arxiu (todos.pir), executaré la comanda aconvert i el transformaré en un arxiu .clw, un aliniament que contindrà tota la informació desitjada (todos.clw). Aquest arxiu todos.clw, mostra lleugeres diferències en el càlcul de lŽestructura per a cada un dels models obtinguts i sobretot cal remarcar que lŽarxiu mostra força dissimilaritats entre el càlcul teòric de lŽestructura secundària i la seva predicció per a cada un dels models:
|